Objectifs
BYTE-Sea coordonne le développe des interfaces permettant d’accéder aux données, de suivre l’avancement et de piloter le programme ATLASea.
1. Interopérabilité et publication des données
Le projet BYTE-Sea garantit l’interopérabilité et la sécurité des données du programme ATLASea, et facilite leur diffusion et leur utilisation conformément aux principes FAIR et Open Science. En collaboration avec le projet SEQ-Sea, il participe à la validation et à la publication des données génomiques dans les entrepôts de référence (European Nucleotide Archive – ENA) : données brutes, génomes assemblés, annotations structurelles et fonctionnelles.
2. Visualisation des données
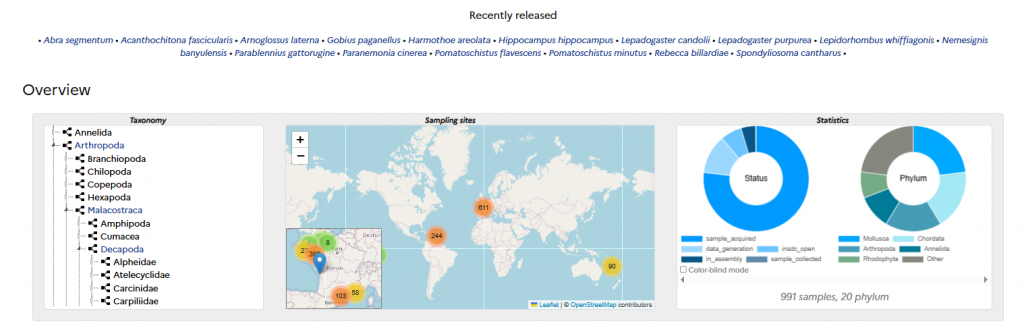
Un portail de données permet de suivre les espèces en cours d’analyse, leur échantillonnage, la production et la publication des données génomiques en lien avec les référentiels de taxonomie et de biodiversité.
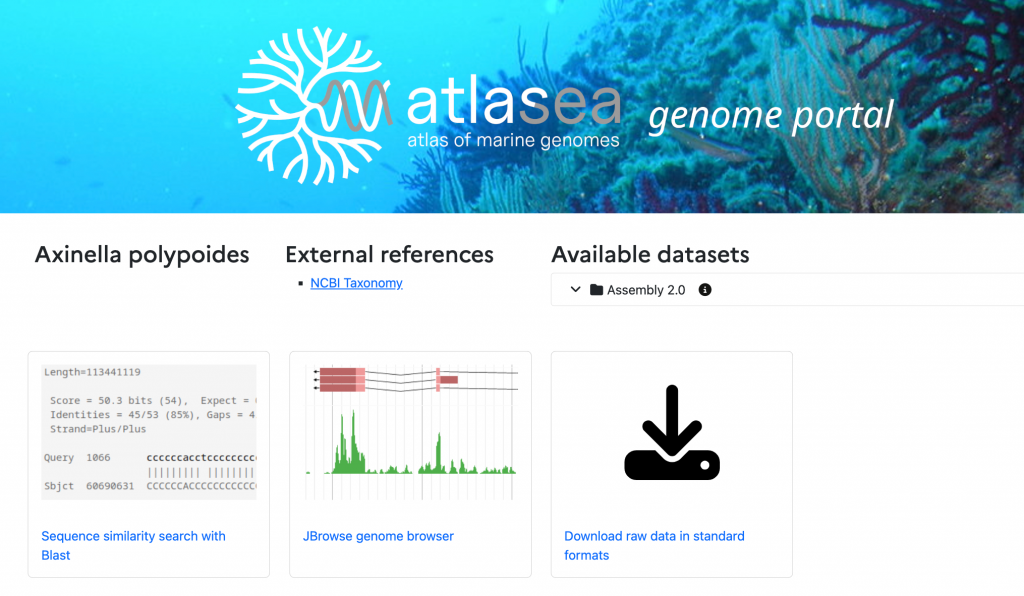
Ce portail donne également accès à des outils de visualisation génomique (Genome Browser) et recherche de similarité, et intégrera prochainement des outils d’analyse de premier niveau (analyse fonctionnelle et comparative).
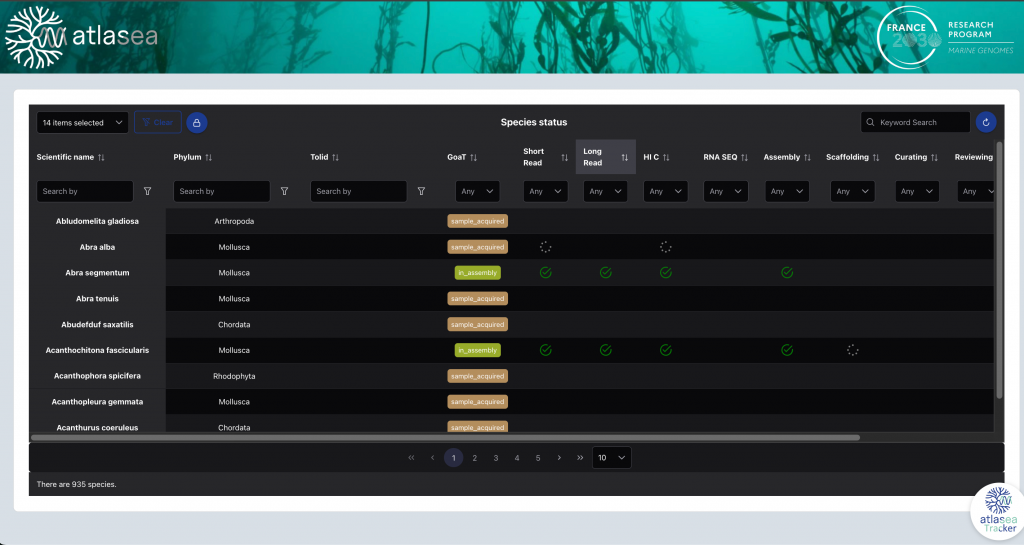
Un portail de suivi, actuellement en cours de développement, dont l’accès est limité aux membres du consortium, permettra de suivre l’évolution du traitement des échantillons, de leur validation taxonomique jusqu’à la publication des assemblages et de l’annotation dans les entrepôts, en passant par les différentes phases de préparation d’ADN et de séquençage.
3. Annotation
La prédiction de la structure et de la fonction des gènes chez les eucaryotes est une tâche complexe qui dépend largement des connaissances préalables et des ressources disponibles pour les espèces concernées. En réponse à ce défi, le programme ATLASea, qui vise à séquencer les génomes d’une large diversité d’espèces pour lesquelles les ressources sont limitées et les proches parents non séquencés, a mis en œuvre une approche méthodique.
Cette approche, en collaboration avec le projet SEQ-Sea, consiste dans un premier temps à automatiser les procédures d’annotation structurales et fonctionnelles. Par la suite, un environnement d’annotation communautaire sera développé afin d’améliorer la prédiction des structures et des fonctions des gènes après leur première publication.
4. Analyse comparative
Un portail permettant d’accéder à l’ensemble des données de génomique d’organismes marins générées par le programme ATLASea et par d’autres consortiums est en cours de développement. L’environnement offrira un accès aux données de séquençage de génomes haute qualité (séquence complète du génome, niveau du chromosome).
Les données mises à disposition incluront des informations sur la séquence génomique, les gènes et les régions codantes et non codantes, les fonctions. Il proposera également des outils pour la recherche de séquences, l’analyse évolutive et fonctionnelle de génomes, ainsi que la visualisation des données dans un contexte environnemental.
Moyens
Les infrastructures numériques principales du projet sont localisées sur le site IFBcore (IDRIS-Orsay) et sur l’infrastructure BigOuest issue de la mutualisation des infrastructures ABiMS (Roscoff) et GenOuest (Rennes).
Nous utilisons les ressources de stockage et de calcul dans les datacenters impliqués qui assurent une disponibilité accrue des données et des portails en répartissant les points d’accès sur trois infrastructures (Orsay, Rennes, Plouzané).
Organisation
Organisme pilote : Institut français de bio-informatique (IFB)
Responsable : Erwan Corre
| Équipe de Station Biologique de Roscoff | Équipe de l’Institut de Biologie de l’École Normale Supérieure (IBENS) | Équipe de l’Institut de Recherche en Informatique et Systèmes Aléatoires (IRISA) | Équipe de l’Institut Français de Bioinformatique (IFB) | Équipe de l’Ifremer |
| Loraine Guegen Mark Hoebeke Annie Lebreton Alexandre Nicaise Gildas Le Corguille | Lucile Jeusset Samuel Lalam Alexandra Louis Hugues Roest Crollius | Anthony Bretaudeau Théo Foulquier Romane Libouban Mateo Boudet | Nicole Charrière Julien Seiler | Pauline Auffret Patrick Durand Laura Leroi Yaëlle Pihan |
Financement
Le programme de recherche exploratoire « ATLASea : Atlas des génomes marins » et le projet ciblé BYTE-Sea (ANR-22-EXAT-0004) bénéficient d’une aide de l’État gérée par l’Agence Nationale de la Recherche (ANR) dans le cadre du plan d’investissement d’avenir « France 2030 ».
